El funcionamiento de los procesos biológicos que tienen lugar en el interior de las células es de una complejidad extraordinaria. Consideremos por ejemplo la apoptosis, el proceso por el cual una célula se «suicida» de manera programada (a diferencia de la necrosis, que tiene lugar de manera incontrolada debido a lesiones en el tejido celular). Se trata de un proceso de extraordinaria importancia, ya que permite que células dañadas o infectadas dejen de consumir recursos, y que sus restos puedan ser reciclados o eliminados de manera segura. Un fallo en el mecanismo de la apoptosis puede desembocar en la aparición de un tumor, debido a la reproducción sin control del tejido. Es por esto que el estudio de los mecanismos que controlan la apotosis es tan importante. La complejidad de esta tarea es sin embargo enorme: hay un gran número de eventos bioquímicos que han de tener lugar de manera orquestada para que se produzca la apoptosis. Podemos imaginarnos un gran diagrama de interacciones en el que una multitud de biomoléculas se interconectan mediante relaciones de activación o de inhibición.
¿Cómo se enfoca el estudio de este tipo de sistemas? De una manera muy experimental, intentando descubrir las relaciones existentes mediante pequeñas alteraciones en los componentes (ahora se desactiva esto, ahora se activa lo otro) y observando los efectos que se producen. Esta metodología ha producido durante los últimos años una media de 10,000 artículos anuales sólo sobre apoptosis, la mayoría de los mismos puramente descriptivos, y frecuentemente contradictorios con otros trabajos. El resultado es que como dice el Prof. David Papermaster, cuantos más datos aprendemos sobre la apoptosis, menos entendemos el proceso. La paradoja puede ser quizás excesiva en sus términos, pero ha motivado una interesante reflexión del Prof. Yuri Lazebnik en un artículo titulado
publicado en Cancer Cell, y luego reimpreso en Biochemistry. Con bastante humor, el Prof. Lazebnik lanza una carga de profundidad contra la metodología que se sigue en el área, imaginando qué pasaría si se siguiera la misma para arreglar una radio. El resultado es realmente hilarante.
Todo comienza por la obtención de una buena provisión de radios en perfecto funcionamiento para poder diseccionarlas y compararlas con la que está rota. Una vez se hubiera descubierto cómo abrirlas, se vería que en su interior habría multitud de objetos de diferentes formas, colores y tamaños, que se intentaría clasificar en familias: los redonditos de dos patitas por allí, los que tienen tres patitas por allá, etc. El primer experimento consistiría en ver si el color afecta en algo a estos objetos. Tras cambiar unos cuantos por otros del mismo tipo pero diferente color, se apreciaría que el sonido de la radio es diferente, atenuado o distorsionado, lo que daría lugar a un gran número de artículos científicos, y a un animado debate.

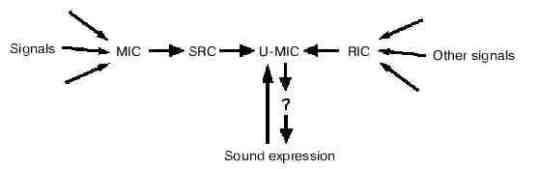
¿Hay algún componente que sea imprescindible para que la radio emita sonido? Tras probar a eliminar diferentes componentes, un postdoc afortunado descubre que hay un cable cuya eliminación hace que cese el sonido. Este cable pasa a denominarse SRC (Componente Recuperado Serendipiedósamente), y se descubre que une a un componente extensible con el resto de la radio. A este componente extensible se le llama MIC (Componente Más Importante), y tras ser estudiado en profundidad se descubre que debe ser de metal, y cuánto más largo mejor (lo que proporciona una explicación evolutiva de por qué es extensible). La importancia de MIC empieza a cuestionarse cuando un estudiante de doctorado descubre que hay una barrita de grafito que también se requiere para que funcione el sistema. A esta barrita se la denomina RIC (Componente Realmente Importante), y comienza la controversia acerca de cuál es más importante, MIC o RIC. Gradualmente se descubre que algunas radios requieren MIC, y otras requieren RIC, lo que anima más el debate, hasta que un astuto postdoc descubre que hay un interruptor que determina si hace falta MIC o RIC para escuchar música. A dicho interruptor se le denomina U-MIC (Componente Indudablemente Más Importante), y su papel central se refleja en diagramas como el que sigue:
Eventualmente se catalogan todos los componentes, y sus interacciones, pero ¿es esto suficiente para arreglar la radio? Si se trata de que uno de los componentes está carbonizado en la radio estropeada, pero no en la radio que funciona, parece claro que la solución es reemplazarlo por uno nuevo. Sin embargo, si el problema es que hay un desajuste entre los componentes de la radio, la probabilidad de que se encuentre la forma de arreglarla es mucho menor, casi imposible según el Prof. Lazebnik. La única solución para él es emplear un lenguaje formal, que evite las típicas vaguedades de los artículos sobre el tema, por ejemplo:
El equilibrio entre proteinas BCL-2 pro- y anti-apoptóticas parece controlar la viabilidad celular, y aparentemente se correlaciona a largo plazo con la habilidad de formar tumores.
El uso de un formalismo haría más evidente la necesidad de medir los parámetros del sistema, y ayudaría a entender su rol. El empleo de un formalismo de este tipo puede ser inevitable en el futuro si se quieren entender los procesos biológicos del mismo modo que entendemos los procesos eléctricos que hacen funcionar una radio. El problema es si los biólogos están preparados (no intelectualmente, que se supone que sí, sino conceptualmente) para ello. Veremos.